Code
nextRender <- (lubridate::now() + lubridate::hours(12)) |>
lubridate::with_tz(tzone = 'Europe/Vienna')2024-04-01T00:02:16+00:00
Hinweis: Die Seite wird circa alle 12 Stunden mit neuen Daten befüllt. Nächstes Update um circa 2024-04-01 14:45:26.
if(!is.null(obsResults)){
distinctObserver <- obsResults$user.id |> unique()
distinctSpecies <- obsResults |>
filter(taxonRank == 'species' | taxonRank == 'subspecies') |>
pull(scientificName) |>
unique()
statsObserver <- obsResults |>
count(user.id) |>
summarise(
mean = round(mean(n), 1),
median = median(n),
)
} else {
distinctObserver <- c()
distinctSpecies <- c()
statsObserver <- tibble(mean = c(0), median = c(0))
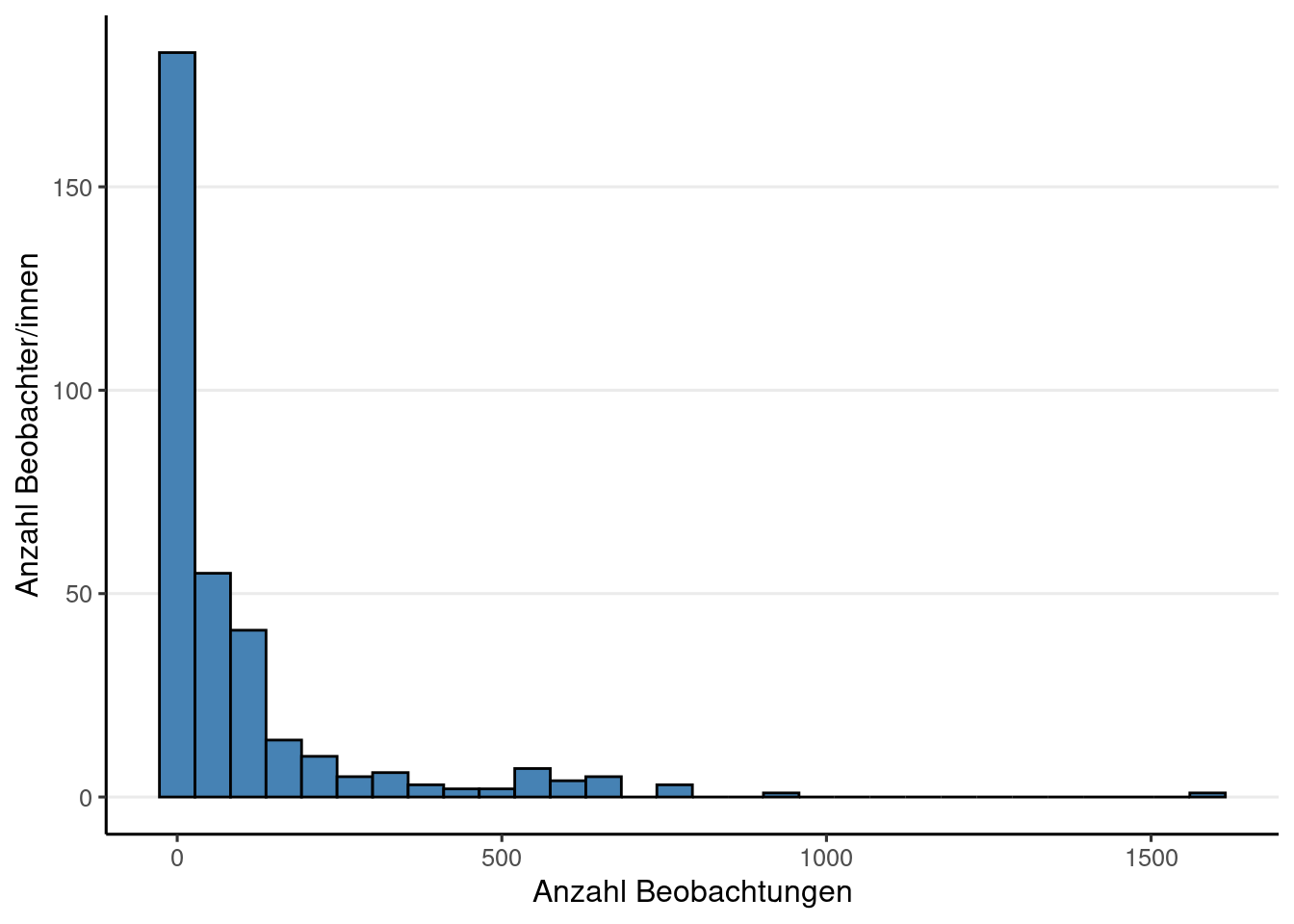
}Insgesamt wurden 33583 Beobachtungen von 342 Beobachterinnen und Beobachtern hochgeladen. Das ergibt einen Mittelwert von 98.2 Beobachtungen pro Beobachter bzw. Beobachterin und einen Median von 21. Auf Spezies-Ebene wurden 3286 Beobachtungen hochgeladen.
if(!is.null(obsResults)){
obsResults |>
count(user.id) |>
ggplot(aes(x = n)) +
geom_histogram(bins = 30, fill = 'steelblue', color = 'black') +
labs(
x = 'Anzahl Beobachtungen',
y = 'Anzahl Beobachter/innen'
) +
scale_y_continuous(
breaks = scales::pretty_breaks(),
) +
scale_x_continuous(
breaks = scales::pretty_breaks(),
) +
theme(
panel.grid.major.y = element_line()
)
} else {
print('Noch keine Beobachtungen')
}
if(!is.null(obsResults)){
tempDf <- obsResults |>
drop_na(time_observed_at)
if(nrow(tempDf) > 0) {
obsResults |>
drop_na(time_observed_at) |>
mutate(
time_observed_at = lubridate::ymd_hms(time_observed_at, tz = "Europe/Vienna", quiet = TRUE),
hour_observed_at = hms::as_hms(time_observed_at),
weekday_observed_at = lubridate::wday(
time_observed_at,
label = TRUE,
week_start = 1,
locale="de_AT"
)
) |>
select(time_observed_at, weekday_observed_at, hour_observed_at) |>
ggplot(aes(x = hour_observed_at, fill = weekday_observed_at)) +
geom_histogram(binwidth = 60*60, color = 'black') +
labs(
x = 'Stunde',
y = 'Anzahl Beobachtungen',
fill = 'Wochentag'
) +
scale_y_continuous(
breaks = scales::pretty_breaks(),
) +
scale_x_time(
breaks = scales::breaks_width("1 hour"),
labels = scales::label_time(format = "%H")
) +
theme(
panel.grid.major.y = element_line()
) +
facet_wrap(~weekday_observed_at, ncol = 1)
}
} else {
print('Noch keine Beobachtungen')
}
if(!is.null(obsResults)){
mapDf <- obsResults |>
drop_na(location, time_observed_at) |>
separate(location, c('latitude', 'longitude'), sep = ',', remove = FALSE, convert = TRUE) |>
mutate(
time_observed_at = lubridate::ymd_hms(time_observed_at, tz = "Europe/Vienna", quiet = TRUE),
label = glue("{user.name} <br/> {scientificName} <br/> {time_observed_at} <br/> <a href='{uri}'>Beobachtung auf iNat</a>"),
group = lubridate::wday(
time_observed_at,
label = TRUE,
week_start = 1,
locale="de_AT"
)
)
mapDfSplit <- split(mapDf, mapDf$group)
m <- leaflet() |> # create map with dataset
setView(lng = 14.12456, lat = 47.59397, zoom = 6) |> # fyi geographic center of austria
addTiles()
for(name in names(mapDfSplit)){
if(nrow(mapDfSplit[[name]]) > 0){
m <- m |>
addCircleMarkers(
data = mapDfSplit[[name]],
lng = ~longitude,
lat = ~latitude,
popup = ~label,
label = ~scientificName,
group = name,
clusterOptions = markerClusterOptions()
)
}
}
m |>
addLayersControl(
overlayGroups = names(mapDfSplit),
options = layersControlOptions(collapsed = FALSE)
)
} else {
print('Noch keine Beobachtungen')
}if(!is.null(obsResults)){
isBirder <- obsResults |>
filter(class=='Aves') |>
count(user.id, user.name) |>
slice_max(n = 1, order_by = n, with_ties = FALSE)
mostDuplicatedObservations <- obsResults |>
filter(taxonRank == 'species' | taxonRank == 'subspecies') |>
count(user.id, user.name, scientificName) |>
slice_max(n = 1, order_by = n, with_ties = FALSE)
longestName <- obsResults |>
filter(taxonRank == 'species' | taxonRank == 'subspecies') |>
mutate(
nameLength = nchar(scientificName)
) |>
slice_max(n = 1, order_by = nameLength, with_ties = FALSE)
} else {
isBirder <- tibble(user.id = c(0), user.name = c(0), n = c(0))
mostDuplicatedObservations <- tibble(user.id = c(0), user.name = c(0), scientificName = c(0), n = c(0))
longestName <- tibble(scientificName = c(0), nameLength = c(0))
}User (auch bekannt als Birder) Christoph Posch hat 531 Vogelbeobachtungen hochgeladen - mehr als jede/r andere. Besonders liebt der User bzw. die Userin Christoph Posch die Art Chloris chloris, die er/sie bereits 257 Mal hochgeladen hat. Das Taxon mit dem längsten Namen ist Asplenium scolopendrium scolopendrium, das 37 Zeichen lang ist.
if(!is.null(obsResults)){
shannonDiversity <- obsResults |>
filter(taxonRank == 'species' | taxonRank == 'subspecies') %>% # need magritter pipe here otherwise cant use dot notation
mutate(N = nrow(.)) |>
count(scientificName, N) |>
mutate(
relFreq = n / sum(N),
logRelFreq = log(relFreq)
) |>
summarise(
shannonDiversity = -sum(relFreq * logRelFreq),
speciesRichness = first(N)
)
} else {
shannonDiversity <- tibble(shannonDiversity = c(0), speciesRichness = c(0))
}Die Biodiversität in einem Ökosystem kann durch verschiedene Indizes in der Ökologie gemessen werden, einer davon ist der Shannon-Index H` (Shannon-Weaver-Index). Dieser Index misst die Diversität von Arten in einem Ökosystem und liegt zwischen 0 und 1. Je höher der Index, desto größer ist die Diversität bzw. je geringer die Chance, noch einmal die gleiche Art zu entdecken.
Als Beispiel können wir mit den iNaturalist Daten und nur Beobachtungen auf zumindest Spezies Niveau den Shannon-Index H` berechnen: 0. Die Anzahl der Arten beträgt 24678.
Weiters können wir mit diesen zwei Kennzahlen den Evenness-Index berechnen, welcher einen Wert von 0 ergibt.
In dieser Liste werden auch doppelte Beobachtungen gezählt, jedoch sollte dies nicht als Tipp betrachtet werden, um an erster Stelle zu stehen.
Die erhobenen Beobachtungen werden erst durch die unerschöpfliche Arbeit der Bestimmer/innen zu wertvollen Daten. An dieser Stelle ein großes Dankeschön an alle Bestimmer/innen ohne euch wäre iNaturalist nicht das, was es heute ist.
# This function could (should) be vectorized to speed up the process
getIdentifierAsVector <- function(identifications, observerId){
if(!is.na(identifications)){
if(identifications == 'list()'){
return(c(""))
}
parsedList <- eval(parse(text=identifications))
identifiers <- parsedList$user |>
as_tibble() |>
filter(id != observerId) |>
mutate(
name = ifelse(is.na(name), login, paste(login, ' (', name, ')', sep = ''))
) |>
pull(name)
if(length(identifiers) == 0){
identifiers <- c("")
}
return(identifiers)
} else {
return(c(""))
}
}
if(!is.null(obsResults)){
obsResults |>
mutate(
identifiers = furrr::future_map2(identifications, user.id, getIdentifierAsVector)
) |>
pull(identifiers) |>
unlist() |>
as_tibble() |>
filter(value != "") |>
count(value) |>
arrange(desc(n)) |>
select('Bestimmungen [#]' = n, 'Bestimmer/In' = value) |>
datatable(rownames = FALSE)
} else {
print('Noch keine Beobachtungen')
}Nur Beobachtungen zumindest auf Spezies-Ebene und Research-Grade werden gezählt.
if(!is.null(obsResults)){
obsResults |>
filter((taxonRank == 'species' | taxonRank == 'subspecies') & quality_grade == 'research') |>
distinct(scientificName, user.id, .keep_all = TRUE) |>
count(user.id, user.name) |>
arrange(desc(n)) |>
select('Uniques [#]' = n, 'User' = user.name) |>
datatable(rownames = FALSE)
} else {
print('Noch keine Beobachtungen')
}In dieser Liste sind die am häufigsten beobachteten Taxa auf Spezies-Ebene aufgeführt, die bereits identifiziert wurden. Es ist jedoch nicht erforderlich, dass diese von einer zweiten Person bestätigt wurden, um den Forschungsstandard (Research-Grade) zu erreichen.
if(!is.null(obsResults)){
obsResults |>
filter(taxonRank == 'species' | taxonRank == 'subspecies') |>
count(scientificName, vernacularName) |>
arrange(desc(n)) |>
select('Beob. [#]' = n, 'Wiss. Name' = scientificName, 'Umgangspr. Name' = vernacularName) |>
datatable(rownames = FALSE)
} else {
print('Noch keine Beobachtungen')
}Besonders erfreulich sind natürlich immer einzigartige Funde, die nur von einer Person in der Challenge hochgeladen wurden. In dieser Liste müssen jedoch alle Spezies von einer zweiten Person bestätigt werden, um den Forschungsstandard (Research-Grade) zu erreichen.
if(!is.null(obsResults)){
obsResults |>
filter((taxonRank == 'species' | taxonRank == 'subspecies') & quality_grade == 'research') |>
add_count(scientificName, vernacularName) |>
filter(n == 1) |>
select('Wiss. Name' = scientificName, 'Umgangspr. Name' = vernacularName, 'User' = user.name, "Link" = uri_html) |>
datatable(
rownames = FALSE,
escape = FALSE,
options = list(
columnDefs = list(list(type = 'html', targets = c(3)
)
)
))
} else {
print('Noch keine Beobachtungen')
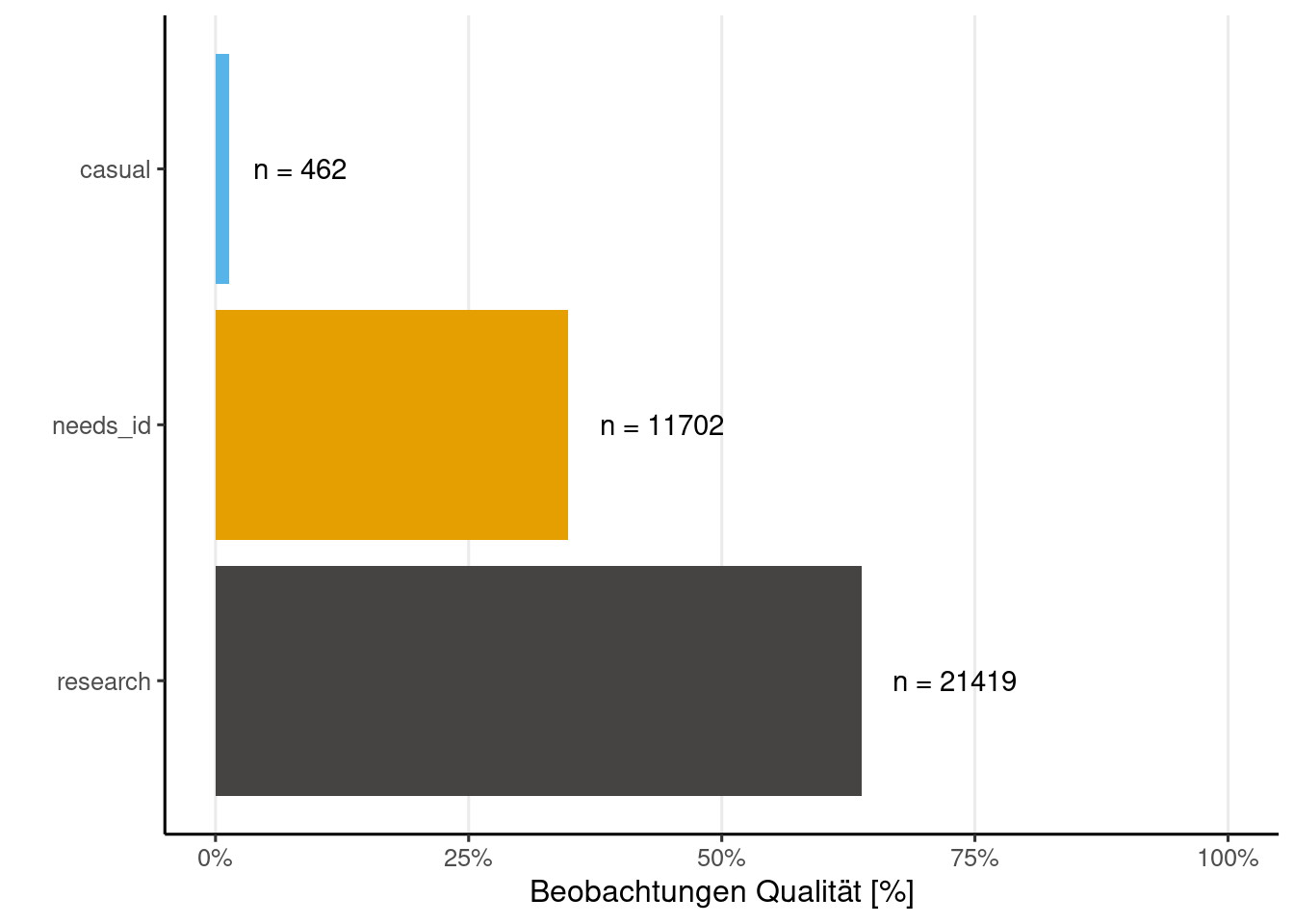
}iNaturalist unterteilt die Beobachtungsqualität in drei Kategorien. Die Kategorie “causal” umfasst Beobachtungen, bei denen mindestens eines der folgenden Elemente fehlt: das Datum der Beobachtung, der Beobachtungsort oder ein Foto (oder Tonaufnahme). Die Kategorie “needs_id” wird angezeigt, bis mindestens zwei Personen eine Spezies für diesen Upload einstimmig bestimmt haben, ohne Widerspruch. Die letzte und wertvollste Kategorie ist “research”, die bestätigte Beobachtungen auf Spezies-Ebene umfasst.
if(!is.null(obsResults)){
obsResults |>
count(quality_grade) |>
arrange(desc(n)) |>
mutate(
np = n / nrow(obsResults),
quality_grade = forcats::fct_reorder(quality_grade, np, .desc = TRUE)
) |>
ggplot() +
aes(x = quality_grade, y = np, fill = quality_grade) +
geom_bar(position = 'dodge', stat='identity', show.legend = FALSE) +
geom_text(aes(label=paste("n =", n)), position=position_dodge(width=0.9), hjust=-0.25) +
labs(
y = "Beobachtungen Qualität [%]",
x = ""
) +
scale_y_continuous(
labels = scales::percent_format(),
limits = c(0, 1)
) +
coord_flip(clip="off") +
theme(
panel.grid.major.x = element_line()
)
} else {
print('Noch keine Beobachtungen')
}
if(!is.null(obsResults)){
obsResults |>
count(phylum) |>
drop_na(phylum) |>
arrange(desc(n)) |>
mutate(
np = n / nrow(obsResults),
phylum = forcats::fct_reorder(phylum, np, .desc = TRUE)
) |>
ggplot() +
aes(x = phylum, y = np, fill = np) +
geom_bar(position = 'dodge', stat='identity', show.legend = FALSE) +
geom_text(aes(label=paste("n =", n)), position=position_dodge(width=0.9), hjust=-0.25) +
labs(
y = "Beobachtungen Phyla [%]",
x = ""
) +
scale_fill_viridis_c(
option = "turbo",
) +
scale_y_continuous(
labels = scales::percent_format(),
limits = c(0, 1)
) +
coord_flip(clip="off") +
theme(
panel.grid.major.x = element_line()
)
} else {
print('Noch keine Beobachtungen')
}
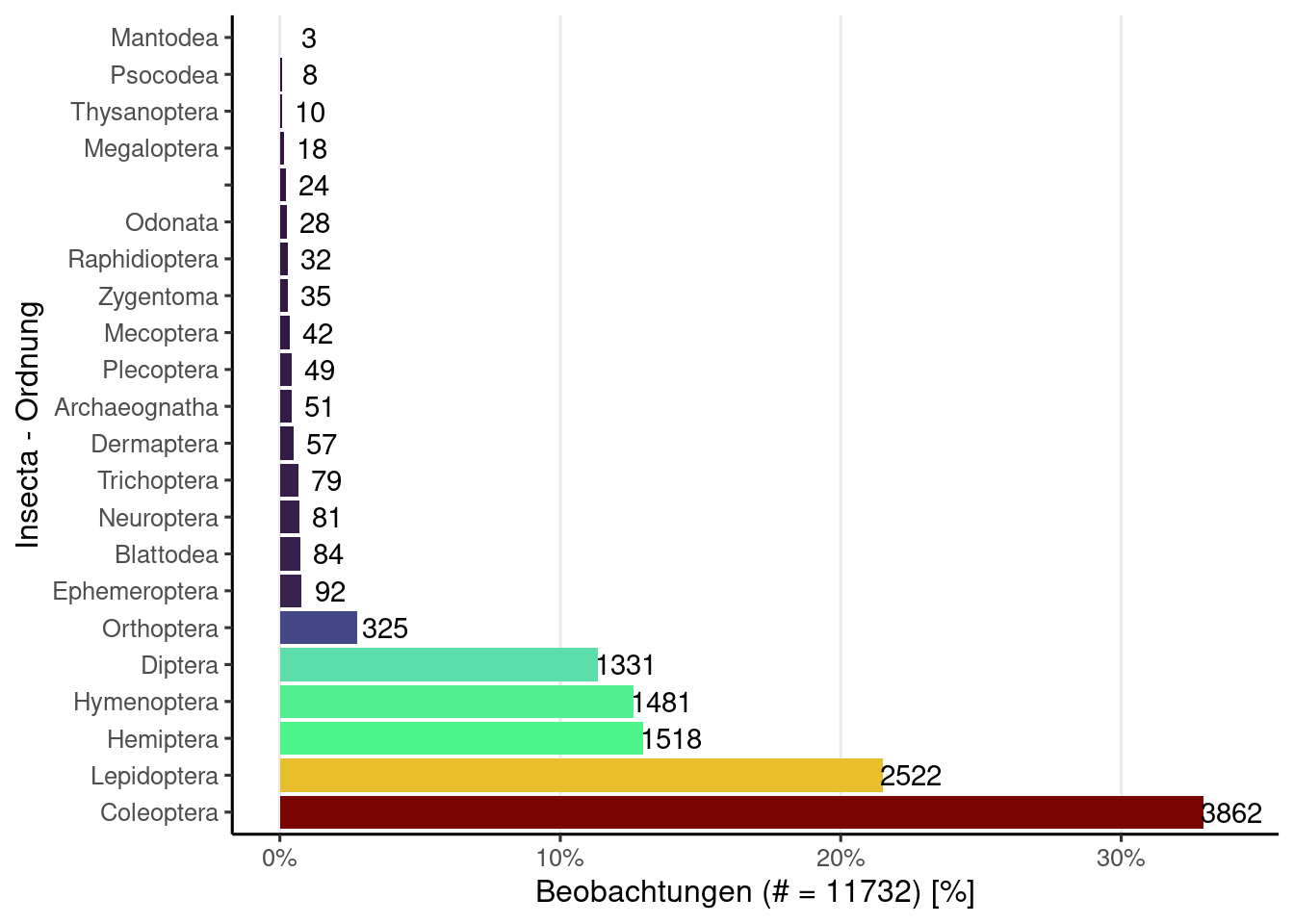
if(!is.null(obsResults)){
obsInsecta <- obsResults |>
filter(class == "Insecta") |>
drop_na(order)
obsInsecta |>
count(order) |>
mutate(
p = n / nrow(obsInsecta),
order = forcats::fct_reorder(order, p, .desc = TRUE)
) |>
ggplot(aes(y = order, x = p, label = n, fill = p)) +
geom_col(show.legend = FALSE) +
geom_text(nudge_x = 0.01, check_overlap = TRUE) +
scale_x_continuous(
breaks = scales::pretty_breaks(),
labels = scales::label_percent()
) +
scale_fill_viridis_c(
option = "turbo",
) +
labs(
y = "Insecta - Ordnung",
x = glue("Beobachtungen (# = {nrow(obsInsecta)}) [%]")
) +
theme(
panel.grid.major.x = element_line()
)
} else {
print('Noch keine Beobachtungen')
}
if(!is.null(obsResults)){
obsResults |>
filter(num_identification_disagreements > 0) |>
select("Link" = uri_html, "User" = user.name, "Taxa" = scientificName) |>
datatable(
rownames = FALSE,
escape = FALSE,
options = list(
columnDefs = list(list(type = 'html', targets = c(1)))
)
)
} else {
print('Noch keine Beobachtungen')
}Anmerkung: Keine Ahnung wie aktuell meine Rote Liste ist.
if(!is.null(obsResults)){
checkList |>
right_join(obsResults, by = c("scientific_name" = "scientificName")) |>
drop_na(annex_II) |>
add_count(scientific_name) |>
count(scientific_name, vernacularName, annex_II_priority, annex_II, annex_IV) |>
arrange(scientific_name) |>
select("Wiss. Name" = scientific_name, "Umgangspr. Name" = vernacularName, annex_II_priority, annex_II, annex_IV, "Beob. (#)" = n) |>
datatable(rownames = FALSE)
} else {
print('Noch keine Beobachtungen')
}if(!is.null(obsResults)){
birdsList |>
right_join(obsResults, by = c("scientific_name" = "scientificName")) |>
drop_na(AnnexI) |>
arrange(scientific_name) |>
count(scientific_name, vernacularName, AnnexI) |>
select("Wiss. Name" = scientific_name, "Umgangspr. Name" = vernacularName, AnnexI, "Beob. (#)" = n) |>
datatable(rownames = FALSE)
} else {
print('Noch keine Beobachtungen')
}